Druck-Version
Faktenwissen
Evolutionsbeweis durch endogene Retroviren
Wie Viren die gemeinsame Abstammung von Mensch u. anderen Primaten beweisen
Wenn wir in der Zeit 10 Millionen Jahre zurückreisen könnten und dort einige Primaten mit Markierungen an verschiedenen Stellen der DNA versehen würden und diese Markierungen heute bei Menschen und Schimpansen wiederfinden würden, dann wäre die gemeinsame Abstammung bewiesen. Zeitreisen sind nicht möglich, aber freundlicherweise haben Viren die Markierungen für uns angebracht.
In diesem Beitrag möchte ich erklären, wie endogene Retroviren die Evolution beweisen oder genauer, die gemeinsame Abstammung von Menschen und anderen Affen. Am besten erkläre ich erstmal, was ein endogenes Retrovirus bzw. ein Virus ist. Ich muss wohl damit anfangen, zu erklären, was eine Zelle tut.
Einführung: Retroviren und Evolution
In jeder Zelle des menschlichen (oder sonst eines) Körpers ist das gesamte Erbgut in den Chromosomen enthalten. Jeder hat bestimmt schon mal davon gehört, dass die DNA dort als Kettenmolekül in Form einer Doppelhelix vorliegt. Die Zelle braucht davon aber nur winzige Abschnitte, nämlich einzelne Gene. Woher die Zelle weiß, welches Gen aus dem langen Strang sie ablesen soll, ist eine interessante Frage und zum Glück für die Argumentation völlig unerheblich.
Die Aufgabe der Zelle ist es, aus den Genen, für die sie zuständig ist, Proteine herzustellen. Das kann so ziemlich alles sein, von Zahnschmelz über Haare bis zu Enzymen. Dazu macht sie von einem DNA-Abschnitt erstmal eine RNA-Kopie: die Boten-RNA oder mRNA. Je drei Buchstaben der mRNA werden dann vom Ribosom in Aminosäuren umgesetzt. Die Aminosäuren werden aneinandergehängt, und die fertige Kette ist dann ein Protein (Abb.1).
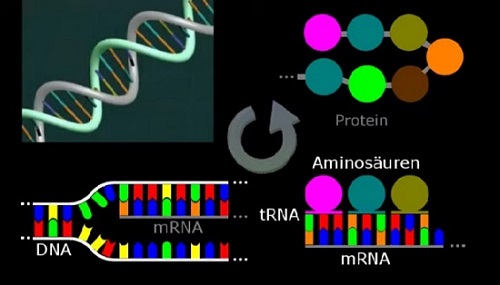
Abb. 1 Einzelne Abschnitte (Gene) auf der DNA werden beim Ablesen in einen komplementären mRNA-Strang umgeschrieben. Jeweils drei "Buchstaben" der mRNA codieren für eine bestimmte Aminosäure, die zu einem Protein verknüpft werden.
Viren gelten nicht als Lebewesen, weil sie für ihre Vermehrung auf Wirtszellen angewiesen sind. Es gibt Viren, die ihre eigene RNA mitbringen und der Zelle als mRNA unterschieben, die dann direkt in Viren-Proteine umgesetzt wird. DNA-Viren lassen ihr Erbgut vorher erstmal von der Zelle in mRNA übersetzen. Die Retroviren setzen noch früher an. Sie enthalten RNA, die von ihrer reversen Transkriptase in DNA umgewandelt und in ein Chromosom der Wirtszelle eingefügt wird. Dieser Schritt verläuft verglichen mit der normalen Verarbeitung rückwärts, daher "Retro"-viren.
Das Aids-Virus HIV ist zum Beispiel ein Retrovirus. Es befällt T-Helferzellen, die für die Immunabwehr wichtig sind. Wenn ein Retrovirus stattdessen Keimzellen befällt und dabei weder die Zelle noch das Wirtstier tötet, kann es passieren, dass die eingefügte virale DNA weitervererbt wird. Man spricht dann von einem "endogenen Retrovirus". Die eingefügte DNA wird "Provirus" genannt, und sie kann bei den Nachkommen des ursprünglich befallenen Tieres zu Viruspartikeln umgesetzt werden. Die Viruspartikel mit Hülle und allem drum und dran nennt man auch "Virion".
So ein endogenes Retrovirus ist nach der Infektion erstmal nur bei einem Individuum vorhanden, und wenn es sich irgendwie schädlich bemerkbar macht, verschwindet es bald wieder durch natürliche Selektion. Wenn es keinen Schaden anrichtet, kann es sich durch genetische Drift in der ganzen Population verbreiten und schließlich fixiert werden. "Fixiert" heißt, dass dann alle Tiere einer Art dieses Provirus in ihrer DNA haben. Es kann auch mehrere hunderttausend Jahre als Allel existieren. Das heißt, dass DNA-Varianten mit und ohne Virus in der Tierart gleichzeitig vorkommen.
Ein Retrovirus hat mindestens die drei Gene gag, pol und env. pol kodiert unter anderem die reverse Transkriptase, und env kodiert die Hülle (Abb. 2). Das Provirus ist an beiden Enden von LTRs begrenzt. LTR steht für "long terminal repeat". Die beiden LTRs sind zum Einfügezeitpunkt identisch aufgebaut, und das in Ableserichtung vordere von ihnen dient als Starter. Es soll also die Zelle dazu bewegen, die Gene abzulesen.
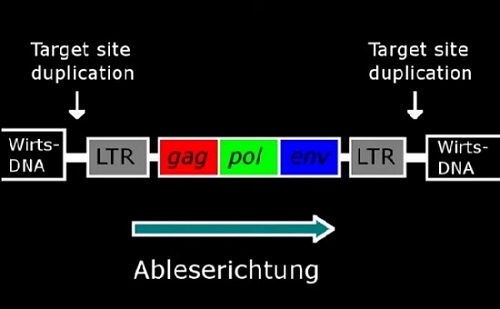
Abb. 2 In die Wirts-DNA eingefügtes Provirus mit den Genen gag, pol und env.
Die Einfügestelle eines Retrovirus in die DNA ist weitgehend zufällig. Es gibt zwar Viren, die genreiche Regionen bevorzugen, aber bei drei Milliarden Basenpaaren - so groß ist die DNA des Menschen - bleiben immer noch Millionen von möglichen Einfügestellen zur Auswahl (Abb. 3). Wenn man Proviren bei verschiedenen Tierarten an einander entsprechenden Stellen findet, kann man also davon ausgehen, dass mal ein Vorfahre beider Arten vom Virus infiziert wurde.
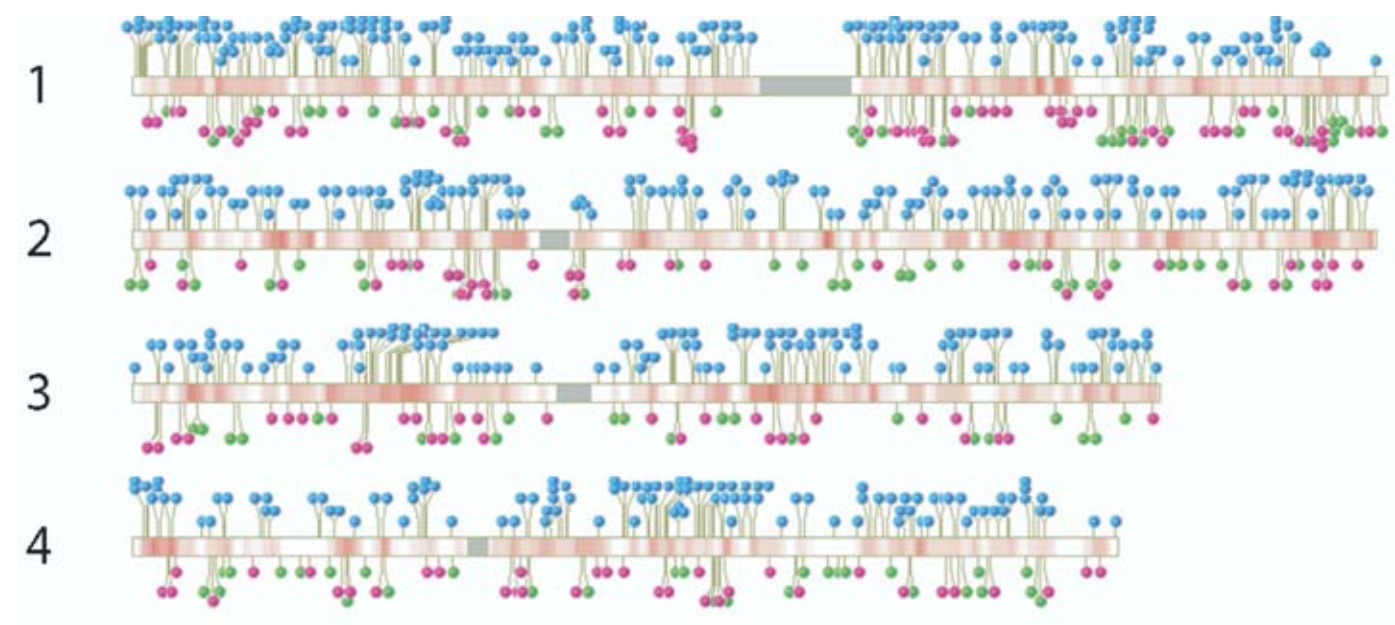
Abb. 3 Die blauen Lollis in diesem Bild stehen für HIV-Einfügestellen. Quelle: MITCHELL et al. (2004)
Der Evolutionsbeweis
Die DNA des Menschen besteht zu etwa 8% aus endogenen Retroviren beziehungsweise ihren Überresten. Oft ist nur noch ein LTR übrig. FINLAY (2006) spricht von 400.000 Einfügestellen.
Vor vier Jahren untersuchten POLAVARAPU et al. (2006) 425 vollständige Retroviren des Schimpansen und fanden 384 davon auch beim Menschen an übereinstimmenden Stellen.
Bei Menschen und Gorillas gibt es weniger Übereinstimmungen und bei Menschen und Rhesusäffchen noch weniger. Wenn man aus der Verteilung von endogenen Retroviren über die Affenarten einen Stammbaum konstruiert, dann entspricht er dem Stammbaum, den man auch aus anderen Merkmalen abgeleitet hat. (Zum Thema "endogene Retroviren als Evolutionsbeleg" s. THEOBALD 2004).
JOHNSON & COFFIN (1999) machten es etwas anders, ihr Evolutionsbeweis beruht auf Proviren, die bei allen untersuchten Affen an der gleichen Position zu finden waren. Dann betrachteten sie dort die Mutationen an den LTRs. Weil beide LTRs zunächst identisch sind, konnten allzu starke Beschädigungen erkannt und in der Statistik als Ausreißer berücksichtigt werden. Aus den mit der Zeit angesammelten und weitervererbten Punktmutationen der LTRs erhielten JOHNSON und COFFIN für jedes einzelne Provirus, das nicht allzu schwere Beschädigungen aufwies, wieder den schon bekannten Stammbaum der Primaten (s. auch HUGHES & COFFIN 2005; Abb. 4).
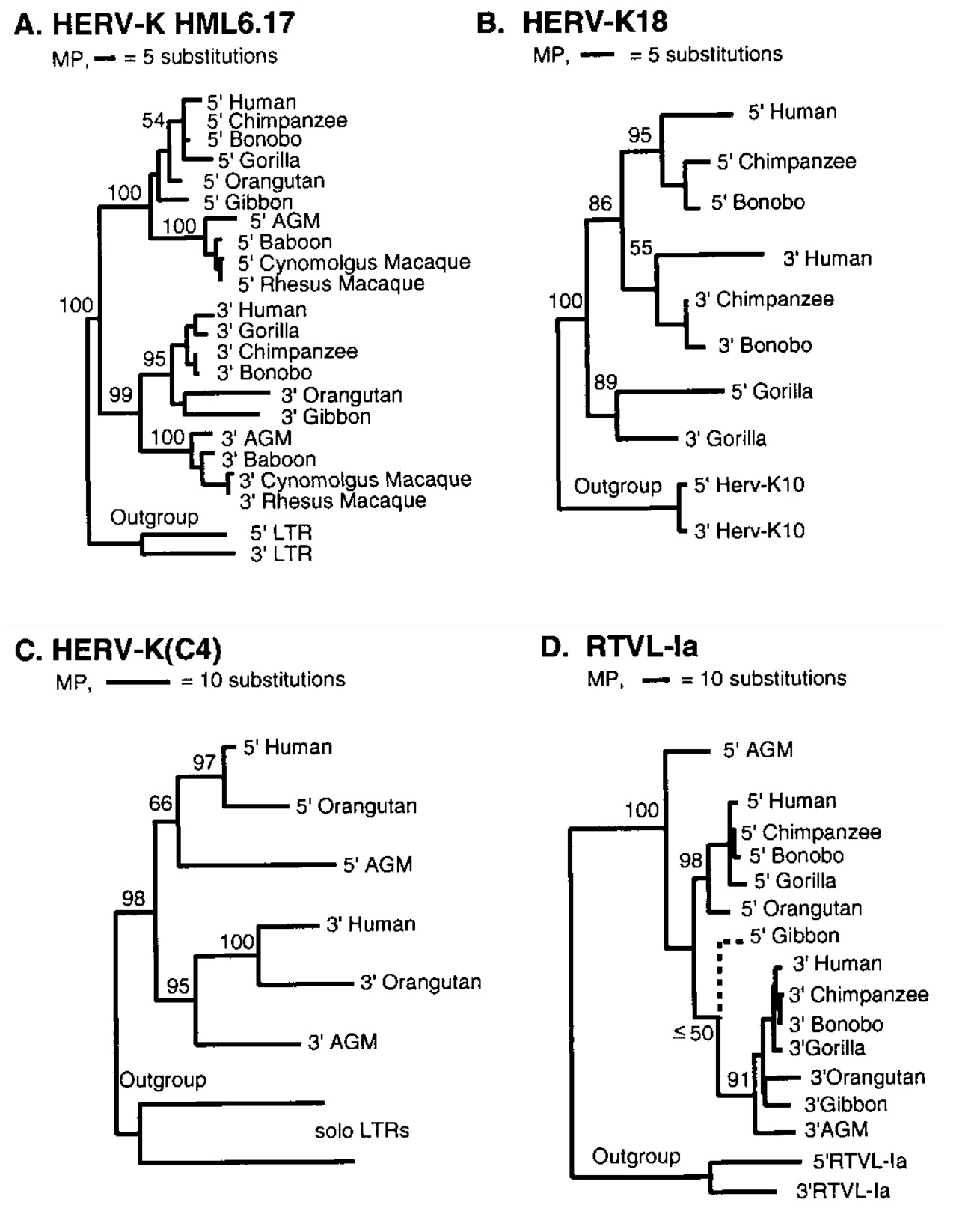
Abb. 4 : Ermittlung der Verwandtschaftsbeziehungen von Primaten anhand verschiedener Proviren. Aus: JOHNSON & COFFIN (1999).
Was sagen die Kreationisten dazu?
Die typische Reaktion ist erstmal zu sagen, dass diese angeblichen Viren in Wahrheit nützliche Bestandteile der ursprünglichen DNA seien. Dazu weisen sie gerne auf die seltenen Fälle hin, in denen ein Gen (und niemals ein ganzes Provirus) eine nützliche Funktion erfüllt. Zum Beispiel wird das Einnisten der Eizelle in die Gebärmutter von einem viralen Gen begünstigt, das die Abstoßung verhindert, was ja für Viren und Eizellen gleichermaßen günstig ist. In anderen Fällen haben LTRs die ursprünglichen Starter von Genen ersetzt. Es sind also ganz selten einzelne Bestandteile von endogenen Retroviren nützlich und niemals das ganze Provirus. Wenn ein ganzes Provirus aktiv ist, dann entstehen tausende von Viruspartikeln, und die Sache kann unangenehm werden.
Neue Einfügungen von endogenen Retroviren, die heute zwar nicht bei Menschen, aber zum Beispiel bei Katzen und Schafen vorkommen, sehen genauso aus wie die alten, aus denen man die Stammbäume ablesen kann, inklusive der so genannten "target site duplications". Es gibt also keine ernsthaften Zweifel, dass die Proviren wirklich von Infektionen herrühren.
Es ist sogar gelungen, aus mehreren Proviren des Menschen eine Retrovirus-Sequenz zu rekonstruieren, die in der Lage war, wieder Viruspartikel zu bilden. Die Forschergruppe um Marie Dewannieux nannte dieses Virus "Phoenix" (DEWANNIEUX et al. 2006).
Der andere Einwand, dass die Einfügestellen nicht zufällig seien, wurde vorhin schon behandelt. Es gibt zwar Vorlieben bestimmter Viren für bestimmte Regionen, aber die erklären keine basenpaargenaue Übereinstimmung. Es wäre außerdem absurd zu glauben, dass die endogenen Retroviren vor Millionen von Jahren immer genau trafen, aber heute nicht mehr. Wenn die Retroviren so gut zielen könnten, warum finden wir sie dann überhaupt an Hunderten von Stellen, aber an diesen Stellen in mehreren Arten, genau dem Stammbaum entsprechend?
Und wieso finden wir nie eine Einfügestelle so verteilt, dass sie dem Stammbaum widerspricht? Etwa, weil die "Evolutionisten" solche Fälle geheim halten? Nein, ein Provirus, das bei Gorillas, Bonobos und Schimpansen vorhanden ist, aber nicht bei Menschen, wurde in den wissenschaftlichen Zeitschriften analysiert (BARBULESCU et al. 2001). Die Erklärung ist, dass das Provirus bei der Abspaltung des Gorillas und auch bei der Trennung von Mensch und Schimpanse noch als Allel vorhanden war und aus der menschlichen Linie verschwand, aber bei den anderen Arten fixiert wurde (Abb. 5).
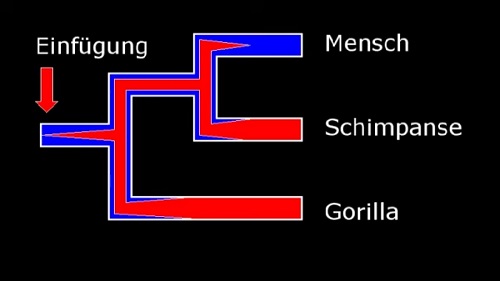
Abb. 5 Die Einfügung eines Provirus beim gemeinsamen Vorfahren von Mensch, Schimpanse und Gorilla. Das als Allel vorhandene Provirus verschwand aus der menschlichen Linie, während es bei den anderen Arten fixiert wurde.
Und wie wollen die Kreationisten erklären, dass sogar die Auswertung der Mutationen an den LTRs immer wieder den bekannten Primaten-Stammbaum ergibt? Vernünftige Erklärungen für die Verteilung der endogenen Retroviren über die Arten habe ich von Kreationisten noch nicht gehört.
Literatur
BARBULESCU, M. et al. (2001) A HERV-K provirus in chimpanzees, bonobos and gorillas, but not humans. Current Biology 11(10), 779-783.
DEWANNIEUX, M. et al. (2006) Identification of an infectious progenitor for the multiple-copy HERV-K human endogenous retroelements. Genome Res. 16(12), 1548-1556.
FINLAY, G. (2006) Human genetics and the image of god.
HUGHES, J. F. & Coffin, J. M. (2005) Human Endogenous Retroviral Elements as Indicators of Ectopic Recombination Events in the Primate Genome. Genetics 171(3), 1183-1194.
JOHNSON, W. E. & COFFIN, J. M. (1999) Constructing primate phylogenies from ancient retrovirus sequences. PNAS 96(18), 10254-10260.
MITCHELL, R. S. et al. (2004) Retroviral DNA integration: ASLV, HIV, and MLV show distinct target site preferences. PloS Biology 2(8), e234.
POLAVARAPU, N. et al. (2006) Identification, characterization and comparative genomics of chimpanzee endogenous retroviruses. Genome Biol. 7(6), R51.
THEOBALD, D. (2004) 29+ evidences for macroevolution. Part 4: the molecular sequence evidence.
Weitere Links zum Thema
Ausführliche Abhandlung im Blog des Autors: Diskussion kreationistischer Einwände
Book Review: 'The Endogenous Retrovirus Handbook' (2022)
Autor: kereng



